Кандидатные генетические маркеры шизофрении на основе данных секвенирования экзома и их связь с иммунологическими, клиническими и морфометрическими изменениями в российской популяции
Цель исследования — поиск генетических маркеров шизофрении на основе данных экзомного секвенирования, а также выявление их потенциальной взаимосвязи с клиническими признаками заболевания, морфологическими изменениями мозга и иммунными нарушениями.
Материалы и методы. Анализируемая выборка состояла из 48 пациентов (23 мужчины и 25 женщин; средний возраст — 31,5±7,7 года) с подтвержденным диагнозом параноидной шизофрении.
Результаты. Выявлены 140 генов с дифференциальными полиморфизмами и обогащенные категории, которые могут быть связаны с патогенезом шизофрении. Анализ генов с диффенциальными частотами функционально значимых распространенных однонуклеотидных полиморфизмов (SNP) по их основным функциям показал, что наиболее часто среди них встречались гены, участвующие в регуляции функций иммунной системы, в развитии нервной системы, а также гены, являющиеся структурными компонентами нейронов и глии, участвующие в восприятии сенсорных стимулов. Полученные данные подтверждают сложность генетической основы шизофрении. Анализ топ 10 генов, содержащих наиболее дифференциальные полиморфизмы, выделяет связанные с шизофренией гены, такие как MUC12 и SH3KBP1. Среди генов, участвующих в регуляции иммунного ответа, одним из наиболее значимо отличающихся по распространенности SNP между группой больных и общей популяцией является ген HLA-DQB2, наличие SNP которого (rs9276572) связано у больных с признаками нарушений функций противовирусного звена иммунитета, со структурными изменениями мозга и нарушениями мышления. Хотя большая часть обнаруженных генов уникальна для исследуемой выборки, для подтверждения их участия в патогенезе данного заболевания и выявления механизмов возникновения и развития болезни необходимы дополнительные исследования. Полиморфизм rs9276572(C) HLA-DQB2 требует дальнейшего изучения как новый потенциальный маркер иммунологических расстройств, морфометрических изменений мозга и нарушений когнитивных функций и при шизофрении. Наши данные указывают на необходимость внедрения персонализированной медицины, так как большинство генетических предпосылок пациент-специфичны и подчеркивают важность дальнейших исследований для понимания генетических аспектов шизофрении и разработки инновационных подходов к ее диагностике и лечению.
Введение
Шизофрения остается одним из наиболее загадочных и сложных психических расстройств, которое влияет на жизнь миллионов людей по всему миру. При достаточно высокой степени наследуемости, которая составляет от 50 до 80%, механизмы развития данного заболевания все еще недостаточно изучены вследствие его мультифакториальности и полигенности [1–4]. В геномных исследованиях последних десятилетий выявлено около 300 распространенных генетических вариантов (через исследования ассоциаций по всему геному) и более 20 редких вариантов (через секвенирование всего экзома и исследования вариантов числа копий), ассоциированных с риском развития шизофрении. Параллельно с этим функциональные геномные и нейробиологические исследования позволили получить подробную информацию о клетках мозга (многочисленных типах нейронов, интернейронов в различных областях мозга) и их изменениях при различных вариантах шизофрении. Взятые вместе, эти результаты отражают сложность механизмов шизофрении, указывая на участие заболевания в патогенезе ансамблей генов (полигенность), а не одного гена [5]. Значимую часть генетического вклада в развитие шизофрении вносят ультраредкие и уникальные аллели, которые затрагивают гены, связанные с развитием нервной системы и другими важными функциями организма [6]. При этом гены c распространенными однонуклеотидными полиморфизмами (SNP) взаимодействуют с генами, содержащими мутации de novo в сети белок-белковых взаимодействий, что предполагает взаимное влияние как распространенных, так и редких вариантов SNP при шизофрении [7]. В свете современных технологических достижений в области генетических исследований использование различных вариантов геномного секвенирования становится ключевым инструментом для более глубокого понимания генетических основ шизофрении [8, 9].
Экзомное секвенирование, представляющее собой анализ экзома — части генома, которая содержит кодирующие белки участки ДНК, — открывает новые возможности для поиска генетических факторов, лежащих в основе шизофрении. При этом учитывается многогранный характер данного расстройства, патогенез которого включает в себя как генетические факторы, так и факторы окружающей среды. Экзомное секвенирование позволяет более полно охватить генетическую картину, идентифицируя варианты генов, предрасполагающих к развитию шизофрении, и выявляя их связь с различными формами болезни [10]. На данный момент принято считать, что патогенез шизофрении связан с редкими генетическими полиморфизмами, которые вносят большой вклад в развитие заболевания, и частыми полиморфизмами с малой величиной эффекта [11].
Данные эпидемиологических и клинических исследований свидетельствуют о том, что у некоторых пациентов с шизофренией наблюдаются ярко выраженные иммунные нарушения и признаки системного воспаления. Достижения в области нейроиммунологии позволили по-новому взглянуть на механизмы, посредством которых иммунная система влияет на развитие и функционирование мозга, породив разнообразные гипотезы о потенциальной роли иммунной системы при шизофрении. В качестве иммунных механизмов, предрасполагающих к заболеванию, рассматривают нейрональные аутоантитела, праймирование микроглии, а также изменение баланса субпопуляций Т-клеток в центральной нервной системе (ЦНС). В развитии и поддержании иммунных расстройств в ЦНС могут играть роль системное воспаление и хронические латентные инфекции [12, 13].
Среди генетических факторов, влияющих на риск шизофрении, целесообразно выделить ряд полиморфизмов генов иммунного ответа. В частности, с шизофренией ассоциированы SNP генов основных провоспалительных и противовоспалительных цитокинов, включая интерлейкины (IL) IL1-β, IL-6, IL-6R, IL-10, IL-17A и фактор некроза опухоли α (TNF-α) [14–16]. Показана связь с шизофренией отдельных SNP аллелей для генов, участвующих в регуляции нейроиммунных взаимодействий, в том числе связь гена нейротрофического фактора мозга (BDNF) с клиническими проявлениями шизофрении [17]. Интерес в отношении иммуногенетической архитектуры шизофрении представляют также работы, исследующие генетическое перекрытие между шизофренией и иммуноопосредованными расстройствами [18].
Эра геномики открывает новые возможности для оценки иммунологической гипотезы шизофрении с использованием крупномасштабных генетических данных. По результатам полногеномных ассоциативных исследований [12] идентифицированы кандидатные регулирующие иммунный ответ гены, связанные с развитием шизофрении. Важно отметить, что среди них имеются гены (CD14, CLU, DPP4, NGF1-A, HSPD1), которые экспрессируются как в иммунных клетках, так и в мозге, что предполагает их двойную роль в иммунитете и функциях ЦНС [12]. Наглядным примером является также ген компонента комплемента C4, который кодирует белок, ответственный за иммунологические механизмы удаления патогенов и за сокращение синапсов (синаптический прунинг) в ЦНС. Ряд полиморфизмов этого гена ассоциирован с риском шизофрении [19].
Для уточнения роли иммуногенетических факторов в формировании клинически значимых особенностей течения шизофрении актуальны определение как распространенных, так и редких полиморфизмов генов иммунного ответа (частота встречаемости которых наиболее сильно различается у больных шизофренией и здоровых лиц) и изучение ассоциаций SNP с иммунологическими нарушениями, клиническими характеристиками и структурными изменениями мозга у больных.
Цель данного исследования — поиск генетических маркеров шизофрении на основе данных экзомного секвенирования, а также выявление их потенциальной взаимосвязи с клиническими признаками заболевания, морфологическими изменениями мозга и иммунными нарушениями.
Данное исследование важно в нескольких аспектах: во-первых, для определения генетических маркеров, которые могут стать инструментом предварительного скрининга лиц с повышенной предрасположенностью к развитию психических заболеваний; во-вторых, для выявления молекулярных механизмов, лежащих в основе заболевания, что необходимо для более глубокого понимания патогенеза шизофрении и разработки эффективных стратегий лечения.
Поскольку исследуемое заболевание зависит от большого числа редких аллелей, то изучение той или иной этнической принадлежности имеет отдельную ценность. Для таких сложных заболеваний, как шизофрения, характерны пациент-специфические особенности патогенеза, при этом каждый случай является важным для понимания общих механизмов, лежащих в основе заболевания. В этом свете статья представляет собой шаг вперед в направлении персонализированной медицины, расширяя возможности своевременного обнаружения болезни и терапевтического вмешательства для тех, кто подвержен риску развития шизофрении. Надеемся, что результаты этого исследования не только обогатят наше понимание генетических аспектов шизофрении, но и создадут новые перспективы для разработки инновационных подходов в диагностике и лечении этого тяжелого психического расстройства.
Материалы и методы
Подбор и описание пациентов. Исследование было одобрено Независимым междисциплинарным комитетом по этической экспертизе клинических исследований (протокол №12 от 14 июля 2017 г.).
Выборка исследования сформирована из 48 пациентов (23 мужчины и 25 женщин; средний возраст — 31,5±7,7 года), повторно госпитализированных в отделения круглосуточного типа Психиатрической клинической больницы №1 им. Н.А. Алексеева Департамента здравоохранения г. Москвы по поводу обострения психотической симптоматики в рамках диагноза параноидной шизофрении (F20.0 по МКБ-10) в период с 2019 по 2021 г. Исследование проведено в соответствии с Хельсинкской декларацией (2013). Все испытуемые дали письменное информированное согласие после полного описания процедур исследования.
Критерии включения в исследование: соответствие психического состояния на момент обследования критериям шизофрении по МКБ-10; информированное согласие на участие в исследовании.
Критерии невключения: тяжелые соматические заболевания в стадии декомпенсации; острые инфекционные заболевания, иммуновоспалительные и аутоиммунные заболевания в фазе обострения; признаки злоупотребления психоактивными веществами и алкоголем; беременность; отказ от участия.
Клинико-динамические показатели (M±σ) участников исследования: длительность заболевания от начала продромальных симптомов — 12,9±6,7 года; длительность заболевания от манифестации — 6,6±5,1 года. Психометрические показатели при поступлении (M±σ): PANSS (шкала позитивных и негативных симптомов шизофрении; общий балл) — 87,26±24,32 балла; PANSS P (тяжесть продуктивной симптоматики) — 20,76±7,40 балла; PANSS N (тяжесть негативной симптоматики) — 25,24±6,84 балла; PANSS G (выраженность других психических нарушений по общей психопатологической шкале) — 39,96±12,77 балла; BFCRS (шкала оценки кататонии Буша–Фрэнсиса) — 4,61±5,06 балла.
Дизайн исследования: кросс-секционное, наблюдательное.
Выделение ДНК. ДНК выделяли из периферической крови больных шизофренией с использованием наборов QIAamp DNA Blood Mini Kit (QIAGEN, США) согласно рекомендациям производителя.
Концентрацию выделенной ДНК измеряли на флуориметре Qubit 2.0 (Thermo Fisher Scientific, США) с использованием набора Qubit dsDNA BR Assay Kit (Thermo Fisher Scientific, США).
Приготовление Illumina библиотек, секвенирование. Библиотеки для экзомного секвенирования получали с использованием набора KAPA HyperExome Kit (Roche, США) в соответствии с руководством пользователя.
150 нг геномной ДНК фрагментировали на приборе Covaris S220 (Thermo Fisher Scientific, США), после чего к концам полученных фрагментов лигировали специфические адаптеры KAPA Universal Adapter при помощи набора KAPA HyberPrep Kit (Roche, США). Далее следовал двухэтапный отбор фрагментов в диапазоне длин 200–350 п.н. с использованием магнитных частиц AMPureXP Beads (Beckman Coulter, США). Полученные фрагменты были амплифицированы с помощью праймеров, специфичных к адаптерам, и гибридизованы с биотинилированными пробами к таргетным регионам KAPA HyberCapture Reagent Kit (Roche, США) в течение 28 ч при температуре 47°С. Биотинилированные гибриды зондов ДНК были выделены и очищены стрептовидиновыми конъюгированными магнитными частицами, проведена вторая амплификация. С целью удаления нецелевых фрагментов амплификации и димеров адаптеров ДНК-библиотеку очищали с использованием магнитных частиц AMPureXP Beads (Beckman Coulter, США). Определение конечной концентрации и валидацию приготовленной библиотеки проводили на флуориметре Qubit (Life Technologies, США) с использованием набора Qubit dsDNA HS Assay Kit (Thermo Fisher Scientific, США) и на биоанализаторе 2100 (Agilent Technologies, США).
Секвенирование библиотеки выполняли на платформе NovaSeq 6000 (Illumina, США) с использованием проточной ячейки S1 и реагентов для секвенирования NovaSeq 5000/6000 S1 Reagent Kit (Illumina, США; 2×150 циклов).
Биоинформационный анализ результатов секвенирования. Произведено картирование нуклеотидных чтений Illumina на референсный геном человека. Для картирования использовали инструмент Bowtie 2 v. 2.4.4 [20]. Создана база индексов для генома сборки GRCh38 (https://www.ensembl.org/Homo_sapiens/Info/Index) с помощью инструмента Bowtie2-build. Разметка геномных последовательностей на гены была выполнена на основе аннотации из базы данных Ensembl [21] (https://ftp.ensembl.org/pub/release-110/gff3/homo_sapiens/Homo_sapiens.GRCh38.110.chr.gff3.gz) с помощью инструмента BEDTools v. 2.30.0 [22]. Картирование проводили с использованием набора параметров — very-sensitive-local. Сжатие, сортировку и индексирование выровненных последовательностей — с помощью пакета SAMtools v. 1.16.1 [23].
Для выявления SNP из выровненных сиквенсов использовали инструмент bcftools v. 1.13-1 [24].
Полученные варианты были аннотированы с использованием инструмента SIFT 4G Annotator (Sorting Intolerant From Tolerant, SIFT) [25]. С помощью SIFT предоставлены функциональные предсказания для обнаруженных полиморфизмов в форме вариантов TOLERATED (безвредные) и DELETERIOUS (вредные). Обычной проблемой для подобного рода исследований является большое количество полиморфизмов (и, соответственно, генов), которые показывают достоверную разницу в частоте аллелей между экспериментальной группой и контролем. Для того чтобы сократить финальное количество генов, мы применили достаточно жесткие условия фильтрации SNP локусов. Для дальнейшей работы нами отобраны только те локусы, которые содержали гомозиготный генотип по аллелю, предсказанному SIFT-алгоритмом как DELETERIOUS. Далее для тех SNP локусов, которые были представлены в базе данных dbSNP (https://www.ncbi.nlm.nih.gov/snp/), определяли популяционные частоты аллелей согласно базе данных частот SNP аллелей для европейской популяции (SAMN10492695) из NCBI (http://ftp.ncbi.nih.gov/snp/population_frequency/latest_release/freq.vcf.gz). Датасет (версия build_id=20201027095038) содержал обобщенные данные 163 190 человек, которые сравнивали с частотами аллелей в нашей выборке из 48 больных шизофренией. Статистическую значимость разницы в частотах аллелей оценивали с помощью функции poisson.test из стандартного набора функций статистической среды программирования R (https://www.r-project.org/). Из тех локусов, которые были предсказаны как NOVEL (нет в dbSNP), отбирали только гомозиготные по аллелю, предсказанному SIFT-алгоритмом как DELETERIOUS, и встречающиеся в нашей выборке не менее чем у 8 (из 48) пациентов с шизофренией.
Для отобранных таким образом локусов с помощью SIFT-программы были определены гены, в которых эти локусы находились. Функциональный анализ и аннотацию GO (Gene Ontology) категорий, а также анализ на обогащение категорий проводили с помощью веб-сервиса DAVID [26].
Определение морфометрических показателей мозга. В исследование морфометрических показателей мозга и иммунологических параметров (соотношение субпопуляций лимфоцитов, уровень цитокинов, хемокинов и факторов роста в сыворотке крови) вошли 29 из 48 пациентов с шизофренией (14 мужчин и 12 женщин; средний возраст — 31,52±2,17 года), прошедших экзомное секвенирование и углубленную оценку клинической симптоматики; и 29 здоровых добровольцев, сопоставимых с больными по возрасту и полу. МРТ-сканирование выполняли на магнитно-резонансном томографе MAGNETOM Verio 3T (Siemens GmbH, Германия). При получении данных была использована 32-канальная катушка для головного мозга. При выполнении морфометрического исследования серого вещества для каждого испытуемого были получены анатомические данные с высоким разрешением на основе Т1-взвешенной последовательности: TR (время между двумя радиочастотными импульсами) — 1900 мс; TE (интервал между радиочастотным импульсом и пиком сигнала (эхо), индуцированного в катушке) — 2,21 мс; 176 срезов; размер воксела — 1×1×1 мм3. Все полученные структурные изображения были проанализированы в программе FreeSurfer.
Определение субпопуляций лимфоцитов. Определение субпопуляций лимфоцитов проводили методом проточной цитофлуориметрии с использованием моноклональных антител. Для окрашивания клеток использовали моноклональные антитела для ТБНК-иммунофенотипирования (Becton Dickinson, США) к антигенам дифференцировки CD3, CD16, CD56, CD45, CD4, CD19, CD25, CD8, CD127, меченым соответственно FITC (изотиоцианат флуоресцеина), РЕ (фикоэритрин), PerCP-Cy™5.5 (комплекс перидинин-хлорофилл протеина с цианином 5.5), PE-Cy™7 (комплекс РЕ с цианином 7), APC (аллофикоцианин), APC-Cy™7 (комплекс APC с цианином 7), Alexa Fluor® 647.
Мультиплексный анализ. Для мультиплексного анализа на этапе пробоподготовки пробирки с гелем и с активатором свертывания SiO2 центрифугировали в течение 20 мин при 2000 об./мин. После центрифугирования полученную сыворотку аликвотировали по 0,6 мл и замораживали при температуре –20°С до исследования. Для проведения мультиплексного анализа по определению сывороточных белков, включая цитокины, хемокины и факторы роста, использовали наборы реагентов производства Merck (США).
Статистическая обработка данных. Статистическую обработку данных иммунологических, морфометрических и клинических исследований проводили с помощью стандартных пакетов прикладных программ Exсel (Microsoft, 2010), Statistica 10.0 (Stat Soft, США). При обработке результатов пользовались стандартными статистическими методами. Для описания данных использовали средние величины (M), стандартное отклонение (σ) и 95% доверительные интервалы. Нормальность распределения оценивали с помощью критерия Шапиро–Уилка. Для оценки различий показателей между группами использовали критерий Стьюдента. Различия считали статистически значимыми при p<0,05.
Результаты
Для всех пациентов было получено более двух с половиной миллиардов парных нуклеотидных чтений длиной по 150 нуклеотидов; размер секвенируемых фрагментов составил 500–700 нуклеотидов. Количество нуклеотидных чтений, произведенных для каждой ДНК-библиотеки, а также процент откартированных нуклеотидных чтений на референсный геном указаны в приложении 1 к данной статье.
После картирования и определения позиций SNP было получено 2 724 996 полиморфных позиций для всех 48 библиотек (позиция определялась как полиморфная, если хотя бы у одного образца обнаруживалось альтернативное состояние SNP). После аннотации нуклеотидных замен с помощью алгоритма SIFT и отбора генотипов с повреждающей заменой осталось 2337 таких SNP-локусов, в которых хотя бы у одного пациента из нашей выборки больных шизофренией был гомозиготный генотип по такому повреждающему аллелю. В результате дифференциального анализа по частотам аллелей между нашей экспериментальной выборкой и общеевропейской популяцией были отобраны 140 генов, содержащих дифференциальные полиморфизмы.
Список генов, отобранных, как описано выше, представлен в приложении 2 к данной статье. Несмотря на то, что была проведена довольно жесткая фильтрация, мы выявили относительно большое количество генов, которые отличаются в нашей выборке. Однако это намного меньше, чем обычно выявляется генов в подобного рода исследованиях.
Если применить слабые критерии фильтрации, например без условия повреждающего характера полиморфизма или гомозиготного генотипа, то таких генов было бы гораздо больше — тысячи, и описать их было бы значительно сложнее.
Один из широко распространенных способов описания списков генов — это так называемый GO-анализ. Он позволяет определять генные категории, которые обогащены в исследуемом списке генов, т.е. представлены в конкретном списке значимо чаще, чем в случайном списке такого же размера. Мы провели такой анализ для генов, выделенных в нашей группе пациентов. Результаты представлены в табл. 1.
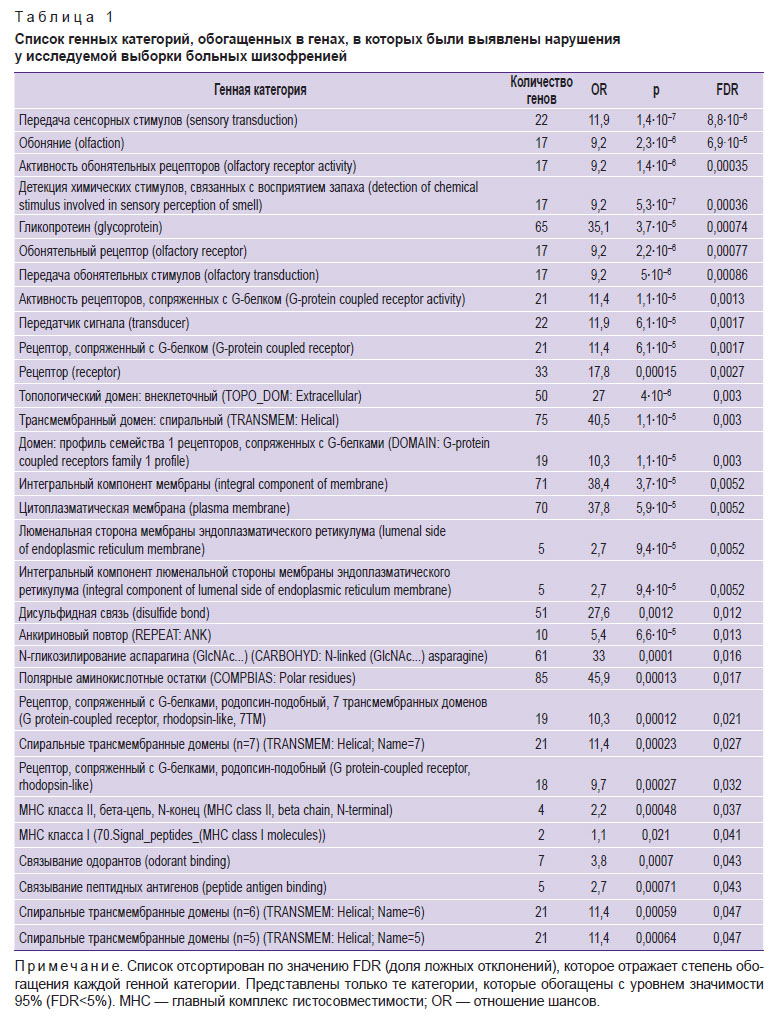
|
Таблица 1. Список генных категорий, обогащенных в генах, в которых были выявлены нарушения у исследуемой выборки больных шизофренией |
Анализ генов с диффенциальными частотами функционально значимых SNP по аннотациям (https://www.genecards.org/) показал, что большую часть этих генов можно объединить в несколько категорий по их основным функциям:
иммунная система — 31;
развитие нервной системы, структурные компоненты нейронов и глии — 29;
сенсорные рецепторы и регуляторы активности сенсорных рецепторов — 19, в том числе 14 обонятельных рецепторов;
транскрипционные факторы — 12;
энергетический метаболизм, митохондриальные белки — 11;
белки микротрубочек, жгутиков и ресничек — 7;
опухолевые антигены — 6;
эпителиальные барьеры — 6;
репарация ДНК и процессинг белков — 3;
функции неизвестны — 4.
Наибольшее число генов с дифференциальной экспрессией относились к регуляторам функций иммунной системы: AGAP1, APOH, ARHGEF40, CANX, CLEC2D, CLEC4M, COPS7A, DDRGK1, GBP3, HLA-B, HLA-DQB2, HSPA1L, ICOSLG, IRS4, KMT2E, MC1R, MMP27, NPLOC4, RNF213, RNFT2, RSRP1, SEMA4D, SH3KBP1, SPINK5, TMEM176B, TNXB, TPSB2, TRIM64C, TSPAN6, UBASH3A.
Анализ генных категорий также показал, что значительное их количество, включая крайне обогащенные, ассоциировано с обонятельными рецепторами. Возможно, существует связь между обонятельными рецепторами и развитием шизофрении: например, через изменения в восприятии окружающей среды и социальных сигналов. Тем не менее данная генная категория часто выделяется при поиске дифференциальных генов и аутлаеров в различных геномных исследованиях. Видимо, это довольно динамичная генетическая категория, подверженная быстрым и легким изменениям. Более того, в связи с использованием строгих критериев фильтрации и отбора поврежденных генов можно утверждать, что гены обонятельных рецепторов в большей степени соответствуют таким условиям, поскольку дефект подобного гена существенным образом не влияет на здоровье современного человека. В то же время многочисленные исследования представили доказательства аномальной обонятельной функции при шизофрении и других психотических расстройствах [27]. Из всех органов чувств обоняние наиболее тесно связано с лобной и височной областями головного мозга; областями, вовлеченными в аффективные и мнемонические функции и патогенез шизофрении. У больных шизофренией были выявлены ухудшение идентификации и различения запахов, изменение порога обонятельной чувствительности, нарушение обонятельной памяти, но на сегодняшний день остаются неясными причины обонятельных нарушений при шизофрении [28].
У мужчин из группы риска развития психоза наблюдалось двустороннее уменьшение объема обонятельной луковицы, аномальная асимметрия задних носовых полостей и обонятельных борозд (левая уменьшалась относительно правой), а также уменьшение объема серого вещества обонятельной коры слева, но не справа. У женщин группы риска развития психоза отмечались схожие изменения, но несколько реже и в меньшей степени. Объем левой обонятельной луковицы у двух групп коррелировал с негативными симптомами [29].
Следует отметить, что обонятельный эпителий играет важную роль в качестве иммунного барьера и участвует в нейроиммунных взаимодействиях [30]. Работ, посвященных генетическим аспектам нарушений обонятельных функций при шизофрении, почти нет, поэтому дальнейшие исследования в этом направлении представляют большой интерес.
На рисунке представлены результаты функционального обогащения по приведенному выше списку генов. Анализ показал, что наибольшую статистическую значимость в этом списке имело обогащение по категории «связывание пептидных антигенов». Связывание антител с пептидными антигенами патогенов необходимо для их нейтрализации и удаления. Кроме того, обращало на себя внимание обогащение по категориям «связывание с лигазой убиквитин-подобных белков», «люминальная сторона мембраны эндоплазматического ретикулума» и «внешняя сторона цитоплазматической мембраны», что указывает на вероятное изменение активности связывания и презентации пептидных антигенов, а также протеасомное расщепление внутриклеточных белков. Результатом этого может являться изменение процессов антигензависимой и антигеннезависимой цитотоксичности, осуществляемой соответственно CD3+CD8+ цитотоксическими T-клетками и CD3–CD16+CD56+ NK-клетками. С учетом того, что ряд генов в списке обладают ингибирующим действием на процессы презентации антигенов и цитотоксичность (CLEC2D, ICOSLG, MC1R, RNFT2, SPINK5, TMEM176B, UBASH3A), можно предположить, что у ряда пациентов с шизофренией их функционально значимые SNP способствуют хронизации инфекций, системному воспалению, а также аутоиммунным реакциям и повреждению собственных клеток.
 |
Результаты функционального обогащения генов, регулирующих иммунные процессы и имеющих дифференциальные частоты функционально значимых SNP у больных шизофренией |
Наиболее статистически значимые различия по частоте функционально значимых SNP с контрольной группой среди генов, участвующих в регуляции функций иммунной системы, отмечались по генам CLEC2D (p=0,00056), IRS4 (p=0,00072), HLA-DQB2 (p=0,0013). Так, у 46,7 и 48% пациентов соответственно встречались в гомозиготном состоянии SNP генов CLEC2D и HLA-DQB2. Первый из них относится к семейству C-лектиновых рецепторов NK-клеток, кодирует белок LLT1, который является лигандом ингибирующего рецептора NK-клеток NKR-P1A. HLA-DQB2 — антиген главного комплекса гистосовместимости II класса, экспрессия которого отсутствует в моноцитах и дендритных клетках, но наблюдается в клетках Лангерганса, где он может участвовать в презентации антигенов CD3+CD4+ T-хелперам [31]. Гены главного комплекса гистосовместимости обладают высокой изменчивостью и кодируют мембранные белки, основные функции которых связаны с представлением антигенных пептидов T-клеткам, регуляцией цитотоксичности лимфоцитов и воспалительного ответа. SNP генов главного комплекса гистосовместимости влияют на риск ряда аутоиммунных и онкологических заболеваний, а также определяют развитие реакции «трансплантат против хозяина» при трансплантации органов. В настоящее время также показана роль белков главного комплекса гистосовместимости в развитии нервной системы и в нейропластичности, в основном за счет регуляции фенотипа клеток микроглии и, в частности, их функции синаптического прунинга (удаления избыточных синапсов для оптимизации функционирования нейросетей), которая может нарушаться при шизофрении [32].
Хотя не доказана связь аллельных вариантов гена HLA-DQB2 с развитием реакции «трансплантат против хозяина», имеется связь уровня экспрессии мРНК и белка HLA-DQB2 с развитием отторжения трансплантата почки [33, 34]. Высокий уровень экспрессии этого гена при раке молочной железы является потенциальным маркером благоприятного прогноза [35].
В табл. 2 представлены результаты оценки параметров клеточного иммунитета; уровня хемокина CXCL10, участвующего в активации Th1-звена иммунитета; иммунорегуляторных белков IL-1RA и TGF-α; фактора роста эндотелия сосудов (VEGF) у больных шизофренией в зависимости от SNP rs9276572(C) гена HLA-DQB2 и в группе здоровых добровольцев. Наличие аллеля G, который встречался у 15 из 29 больных, прошедших иммунологические исследования и исследования морфометрических показателей мозга (всего аллель G был обнаружен у 20 из 48 больных шизофренией, включенных в исследование), было связано при шизофрении с изменениями клеточного иммунитета, включая повышение содержания CD3+ T-клеток, CD3+CD8+ цитотоксических T-клеток, снижение количества CD3–CD16+CD56+ NK-клеток (естественных киллеров). CD3+CD8+ T-клетки и CD3–CD16+CD56+ NK-клетки являются эффекторными цитотоксическими клетками, играющими ключевую роль в удалении зараженных вирусами и измененных клеток организма. Функции CD3+CD8+ T-клеток являются антигензависимыми, а CD3–CD16+CD56+ NK-клеток — антигеннезависимыми.
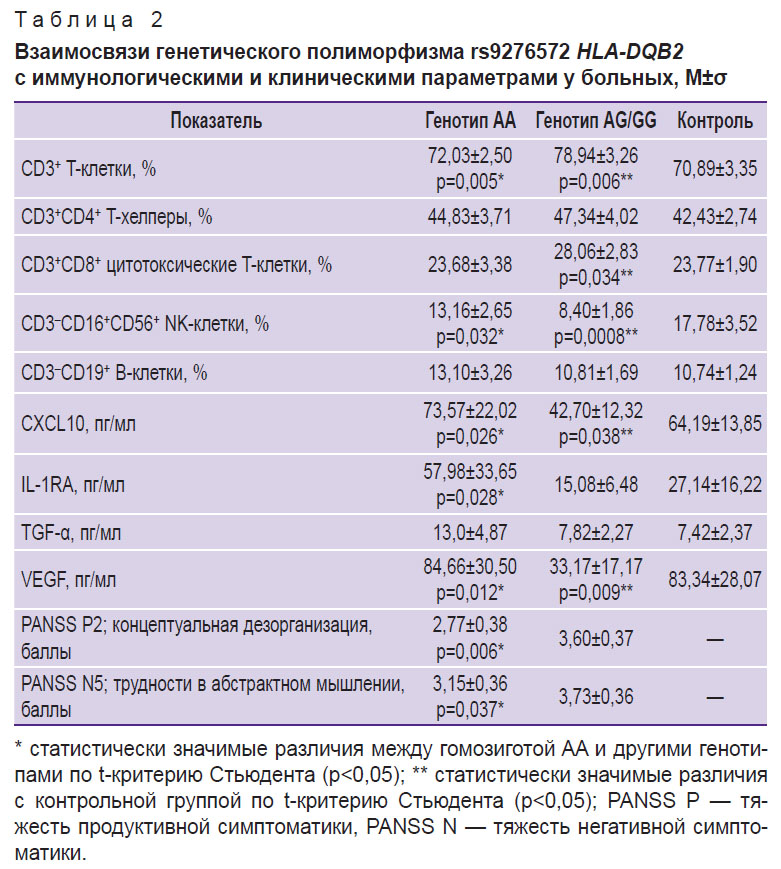
|
Таблица 2. Взаимосвязи генетического полиморфизма rs9276572 HLA-DQB2 с иммунологическими и клиническими параметрами у больных, M±σ |
Кроме того, у больных, имеющих аллель G, было снижено содержание хемокина CXCL10, стимулирующего реакции цитотоксичности. Таким образом, у этих больных имели место признаки нарушений активности механизмов антигензависимой и антигеннезависимой цитотоксичности. У них отмечалось также снижение содержания VEGF, что может указывать на эндотелиальную дисфункцию. VEGF играет важную роль в качестве нейропротекторного фактора, уменьшающего повреждающие эффекты нейровоспаления. В исследованиях invitro показано, что VEGF защищает нейроны от окислительного стресса, гипоксии, гипогликемии и эксайтотоксичности, опосредованной глутаматом [36]. Кроме того, VEGF поддерживает синаптическую пластичность и способствует росту, выживанию, дифференцировке и миграции нейрональных и глиальных клеток, участвует в регуляции кровотока и ангиогенеза в ЦНС. Имеются данные об обратной корреляции между уровнем VEGF и объемом лобной доли мозга при шизофрении, поэтому можно предполагать защитный эффект этого белка по отношению к морфометрическим изменениям мозга у больных [37].
Аллель G SNP rs9276572(C) гена HLA-DQB2 по данным оценки морфометрических показателей мозга ассоциирован с выраженным снижением толщины коры в областях височной и лобной доли мозга, а также в области шпорной борозды затылочной доли (табл. 3). В то же время у пациентов, не имевших этого аллеля, не отмечалось значимого (p<0,01) снижения толщины коры по сравнению с контрольной группой.
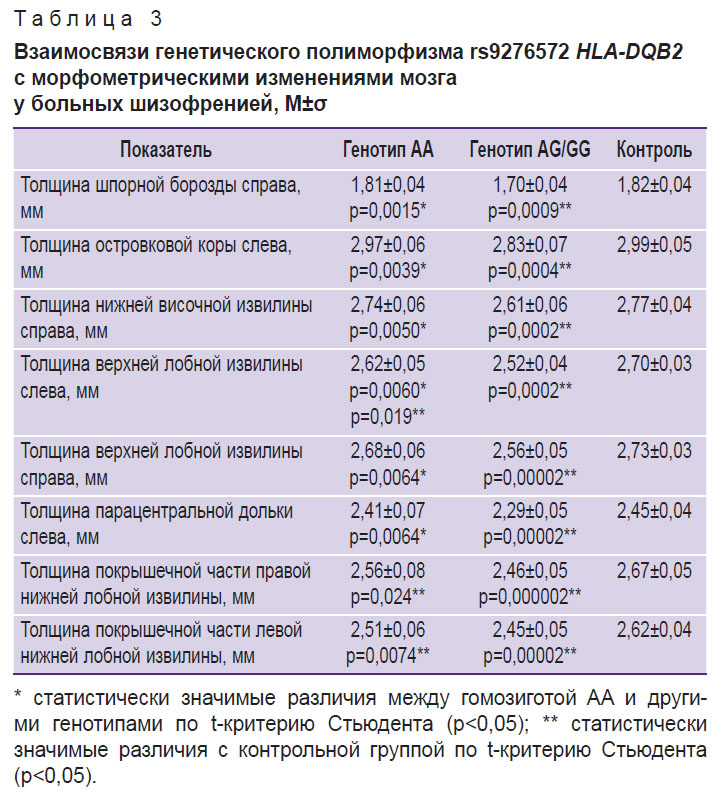
|
Таблица 3. Взаимосвязи генетического полиморфизма rs9276572 HLA-DQB2 с морфометрическими изменениями мозга у больных шизофренией, M±σ |
У некоторых пациентов с шизофренией (6%) также отмечались функционально значимые полиморфизмы гена IRS4, который участвует в передаче сигнала рецепторов с тирозинкиназной активностью, включая рецепторы инсулина; стимулирует митогенные эффекты рецептора IGFR1; регулирует пластический обмен и метаболизм глюкозы. Следует отметить, что инсулиноподобный фактор роста (IGF-1), основной агонист IGFR1, участвует в регуляции нейрогенеза, синаптогенеза, миелинизации аксонов и ветвления дендритов. Ранее нами показано [38], что уровень IGF-1 ассоциирован с выраженностью иммуновоспалительных нарушений, морфометрических нарушений мозга и моторных расстройств (кататонии) при шизофрении. Имеются данные, что уровень IGF-1 связан с хорошей чувствительностью к лечению при шизофрении [39]. Отметим, что в список генов с различиями по частоте функционально значимых SNP вошли в общей сложности 11 генов, регулирующих энергетический метаболизм. С учетом полученных данных можно предположить роль генетически обусловленных нарушений функций митохондрий, энергетического обмена и инсулинового сигналинга, а также иммунных изменений, связанных с нарушением иммунорегуляторных функций IGF-1, в патогенезе части случаев шизофрении.
Еще один способ проанализировать данные NGS — это выбрать некоторое количество генов, которые являются наиболее дифференциальными для данной конкретной выборки пациентов. Нами были выбраны топ 10 генов, разница в частотах аллелей для которых максимальна между пациентами и общеевропейской популяцией. Список этих генов представлен в табл. 4.
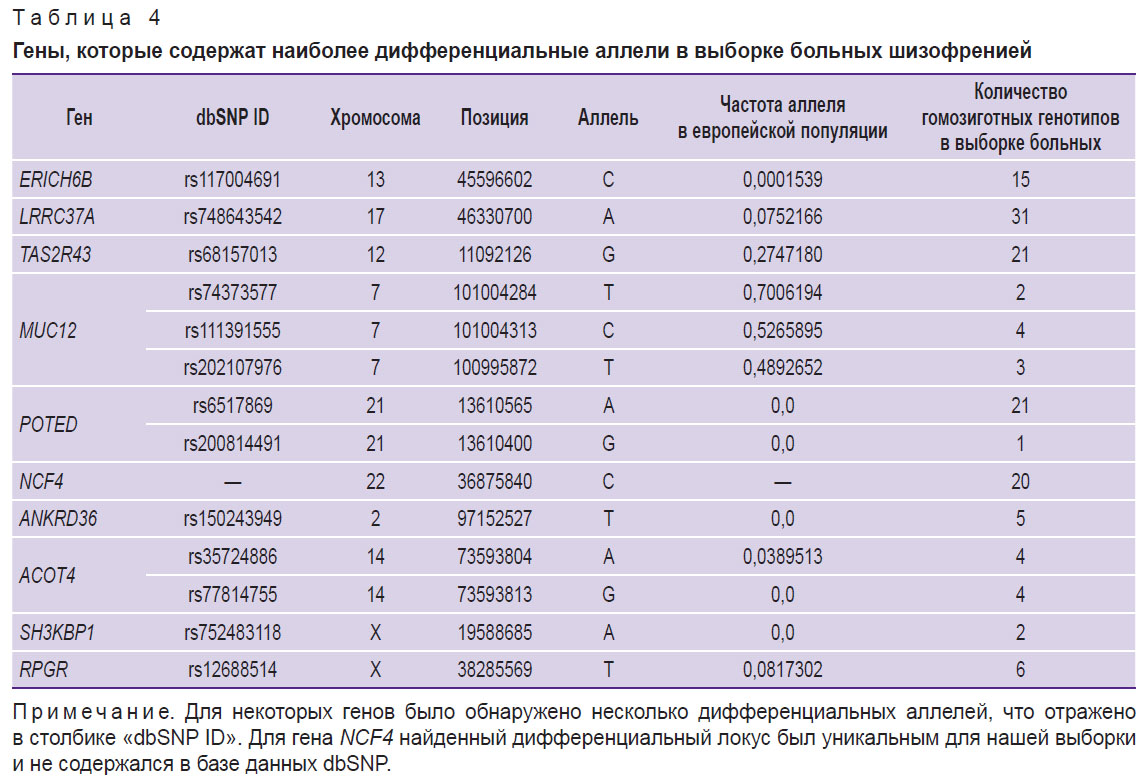
|
Таблица 4. Гены, которые содержат наиболее дифференциальные аллели в выборке больных шизофренией |
Аллели SNP, представленные в табл. 4, классифицируются как повреждающие для соответствующего гена согласно алгоритму SIFT. В большинстве генов отмечается повышенная частота указанного повреждающего аллеля среди лиц, страдающих шизофренией, по сравнению с общеевропейской популяцией. Однако в случае гена MUC12 наблюдается обратная тенденция: частоты «повреждающего» аллеля в популяции достаточно значительны и составляют 0,7 (rs74373577), 0,52 (rs111391555) и 0,48 (rs202107976) для различных позиций в гене. В анализируемой выборке гомозиготным генотипом по данному аллелю обладают всего 2, 4 и 3 человека (из 48 пациентов) соответственно. Важно учесть, что для вычисления теоретической частоты гомозиготного генотипа необходимо возвести частоту аллеля в квадрат.
Существует несколько возможных объяснений данного феномена, таких как снижение предрасположенности к шизофрении в результате поломки соответствующего гена или возможная ошибочность SIFT-алгоритма относительно данного аллеля, который, по всей вероятности, не нарушает функциональность гена MUC12. Однако мы предполагаем, что наиболее вероятным объяснением является наличие дефектной копии гена MUC12 в референсной последовательности, случайно включенной в геномную сборку GRCh38. И поскольку альтернативные аллели гена MUC12, представленные в табл. 4 в столбце «аллель», приводят к значительным изменениям в структуре белка (видимо, к более правильной), алгоритм интерпретирует это как изменение/повреждение. Косвенную поддержку такой гипотезе обеспечивает тот факт, что в данной позиции в белке MUC12 у других видов (например, мыши) обнаружены альтернативные аминокислоты (отличные от человеческих), в то время как окружающие аминокислоты в значительной степени совпадают.
Обсуждение
На данный момент существует несколько онлайн-ресурсов, которые содержат гены-кандидаты, участвующие в патогенезе шизофрении [40–42]. Например, база данных SZDB (http://szdb.org/) [42] содержит в том числе и гены, которые были найдены при анализе экзомного секвенирования выборок больных шизофренией. Количество генов, содержащихся в данной базе, равно 9366, что составляет более трети всех белок-кодирующих генов человека. Это число основано лишь на 16 исследованиях экзомного секвенирования пациентов с шизофренией, хотя таких работ выходит все больше и больше.
Количество выявленных генов говорит о том, что каждая выборка больных содержит большое количество уникальных полиморфизмов, которые вносят вклад в развитие шизофрении у данной группы пациентов. Возможно, почти любой ген может содержать такой аллельный вариант, который в ту или иную сторону будет влиять на предрасположенность к шизофрении или другим сложным заболеваниям. Это может означать, что любое исследование, которое связано с поиском генетических вариантов, влияющих на предрасположенность к шизофрении, имеет важное значение для понимания механизмов, приводящих к заболеванию. В результате накопления такого рода информации должно прийти понимание того, какие генетические изменения наиболее важны для патогенеза шизофрении.
Каждое новое исследование ведет к накоплению знаний и вносит значительный вклад в расширение доступной информации, способствующей выявлению молекулярных причин заболевания. В этом плане наше исследование также полезно для изучения шизофрении и выявления генов, связанных с болезнью. Из 140 генов, обнаруженных в данном исследовании, только 102 есть в базе данных SZDB, остальные гены являются уникальными для нашей выборки больных. Кроме того, из топ 10 генов лишь два — TAS2R43 и NCF4 — присутствуют в SZDB. Это подтверждает идею о том, что у каждого пациента генетические причины болезни индивидуальны, поэтому что-то общее в механизмах предрасположенности нужно искать на верхних уровнях реализации генетического материала.
Мы применили жесткие критерии фильтрации к полученным данным. С одной стороны, это позволило значительно сократить список генов, с которым придется работать, и тем самым упростить дальнейшую оценку результатов, а с другой, ограничило возможности для таких видов анализа, как обогащение GO-категорий и анализ функциональных аннотаций, для которого подходит большое количество генов. В результате анализа функциональных аннотаций помимо категорий, связанных с обонятельными рецепторами (которые, по-видимому, могут быть ассоциированы с заболеванием), мы нашли лишь несколько дополнительных категорий, обогащенных в генном списке больных. К таким категориям относятся люминальная сторона мембраны эндоплазматического ретикулума, интегральный компонент люминальной стороны мембраны эндоплазматического ретикулума, главный комплекс гистосовместимости класса I, связывание пептидных антитенов. Эти категории содержат гены, связанные с презентацией антигенов и иммунным ответом. В нашем списке представлены шесть таких генов: HLA-DRB5, CANX, HLA-B, HLA-C, HLA-DQB2, CLEC4M. На примере большого количества работ продемонстрировано участие этих генов в патогенезе шизофрении и других психических заболеваний. Например, в недавних исследованиях [43–46] было показано, что эти гены связаны с предрасположенностью к шизофрении и с реакцией на антипсихотические препараты.
Аннотирование функций генов, полиморфизмы которых встречались с дифференциальной частотой при шизофрении, позволило выделить значительно большее число генов, связанных с функциями иммунной системы, — более 30. Функциональное обогащение этих генов указывает на изменение активности процессов, связанных с презентацией антигенов, системой антигензависимой и антигеннезависимой цитотоксичности. Нарушение презентации антигенов макрофагами периваскулярных пространств и клетками микроглии ЦНС может являться одним из патогенетических механизмов шизофрении, способствующим развитию и поддержанию хронического нейровоспаления и повреждению нейронов. Это нарушение, возможно, обусловлено комбинированным действием нескольких факторов, включая латентные вирусные инфекции и измененную реактивность микроглии в связи с избыточной иммунной активацией на ранних этапах развития, а также генетической предрасположенностью. Последняя реализуется в том числе за счет изменчивости генов главного комплекса гистосовместимости, обеспечивающих презентацию антигенов и влияющих на регуляцию функций NK-клеток и CD3+CD8+ цитотоксических лимфоцитов. В настоящее время получены убедительные доказательства, что аллельные варианты генов HLA, включая HLA-DQB1*0602 и HLA-DRB1*04, способны влиять на риск шизофрении [12]. Однако связь аллелей гена HLA-DQB2 с шизофренией ранее не изучалась. В данной работе впервые показано, что SNP rs9276572(C) этого гена встречается при шизофрении с повышенной частотой и ассоциирован с нарушениями системы цитотоксичности. При этом количество NK-клеток, осуществляющих антигеннезависимую цитотоксичность, а также уровень хемокина CXCL10, стимулирующего хемотаксис цитотоксических лимфоцитов и NK-клеток, были существенно снижены. В анализируемой группе больных также наблюдалось снижение уровня VEGF, регулирующего ангиогенез в ЦНС и оказывающего нейропротекторные функции. По данным структурной МРТ, эти пациенты имели выраженное снижение толщины коры в зонах лобной и височной доли мозга по сравнению с пациентами, не имевшими этого аллеля, и с контрольной группой. С клинической точки зрения носители аллеля G в SNP-локусе rs9276572 гена HLA-DQB2 характеризовались большей выраженностью расстройств мышления по данным шкалы PANSS (PANSS P2 — концептуальная дезорганизация, PANSS N5 — трудности в абстрактном мышлении).
Кроме того, в нашем генном списке есть несколько обогащенных категорий, связанных с анкиринами. Анкирины представляют собой широко экспрессируемые адаптеры, которые координируют мембранные белки в специализированные домены и связывают их с субмембранным цитоскелетом. Показана роль анкиринов в организации начального сегмента аксона и узлов Ранвье в нейронах. Выявлено участие анкиринов в синапсах, где они не только организуют и стабилизируют рецепторы нейромедиаторов, но также модулируют морфологию дендритных отростков и регулируют адгезию к пресинаптическому участку. Гены анкирина тесно связаны с патологиями нервной системы и психиатрическими расстройствами, включая биполярное расстройство, шизофрению и аутизм [47].
Анализ топ 10 генов, которые содержали наиболее дифференциальные SNP-локусы в нашей выборке больных, показал, что кроме двух генов, представленных в базе SZDB, еще два гена описаны как связанные с шизофренией. В работе [48] ген MUC12 вошел в топ 10 мутированных генов при исследовании случаев ранней шизофрении (childhood-onset schizophrenia). В работе [49] на крысиной модели показано, что ген SH3KBP1 играет важную роль в механизмах возникновения шизофрении, которые связаны с пренатальным дефицитом питательных веществ у матери во время беременности.
Заключение
Наша работа, проведенная с использованием методов экзомного секвенирования, подчеркивает важность данного метода для выявления новых генов, связанных с шизофренией. Большинство выявленных генов являются уникальными для нашей выборки и требуют дополнительного изучения и объяснения. Полиморфизм rs9276572(C) HLA-DQB2 нуждается в дальнейших исследованиях как новый потенциальный маркер иммунологических расстройств, морфометрических изменений мозга и нарушений когнитивных функций и при шизофрении.
В целом наша работа вносит важный вклад в понимание генетических основ шизофрении и подчеркивает сложность и многообразие факторов, влияющих на это расстройство. Дальнейшие исследования в данной области имеют потенциал для разработки более эффективных методов диагностики и терапии, а также для более глубокого понимания биологических основ психических заболеваний.
Источники финансирования. Выбор, клиническое исследование больных, мультиплексный анализ, МРТ-сканирование, выделение ДНК, приготовление библиотек и секвенирование было финансировано проектом Российского научного фонда №20-15-00299П (https://rscf.ru/project/20-15-00299/).
Биоинформационный анализ результатов секвенирования выполнен в рамках государственного задания №123021500064-0.
Определение параметров клеточного иммунитета проведено в рамках выполнения государственного задания НИЦ «Курчатовский институт».
Работа была выполнена с использованием оборудования центра коллективного пользования «Комплекс моделирования и обработки данных исследовательских установок мега-класса» НИЦ «Курчатовский институт» (http://ckp.nrcki.ru/).
Конфликт интересов. Конфликт интересов, связанный с данным исследованием, отсутствует.
Литература
- Owen M.J., Sawa A., Mortensen P.B. Schizophrenia. Lancet 2016; 388(10039): 86–97, https://doi.org/10.1016/S0140-6736(15)01121-6.
- Schizophrenia Working Group of the Psychiatric Genomics Consortium Biological insights from 108 schizophrenia-associated genetic loci. Nature 2014; 511(7510): 421–427, https://doi.org/10.1038/nature13595.
- Pardiñas A.F., Holmans P., Pocklington A.J., Escott-Price V., Ripke S., Carrera N., Legge S.E., Bishop S., Cameron D., Hamshere M.L., Han J., Hubbard L., Lynham A., Mantripragada K., Rees E., MacCabe J.H., McCarroll S.A., Baune B.T., Breen G., Byrne E.M., Dannlowski U., Eley T.C., Hayward C., Martin N.G., McIntosh A.M., Plomin R., Porteous D.J., Wray N.R., Caballero A., Geschwind D.H., Huckins L.M., Ruderfer D.M., Santiago E., Sklar P., Stahl E.A., Won H., Agerbo E., Als T.D., Andreassen O.A., Bækvad-Hansen M., Mortensen P.B., Pedersen C.B., Børglum A.D., Bybjerg-Grauholm J., Djurovic S., Durmishi N., Pedersen M.G., Golimbet V., Grove J., Hougaard D.M., Mattheisen M., Molden E., Mors O., Nordentoft M., Pejovic-Milovancevic M., Sigurdsson E., Silagadze T., Hansen C.S., Stefansson K., Stefansson H., Steinberg S., Tosato S., Werge T; GERAD1 Consortium; CRESTAR Consortium; Collier D.A., Rujescu D., Kirov G., Owen M.J., O’Donovan M.C., Walters J.T.R. Common schizophrenia alleles are enriched in mutation-intolerant genes and in regions under strong background selection. Nat Genet 2018; 50(3): 381–389, https://doi.org/10.1038/s41588-018-0059-2.
- Trubetskoy V., Pardiñas A.F., Qi T., Panagiotaropoulou G., Awasthi S., Bigdeli T.B., Bryois J., Chen C.Y., Dennison C.A., Hall L.S., Lam M., Watanabe K., Frei O., Ge T., Harwood J.C., Koopmans F., Magnusson S., Richards A.L., Sidorenko J., Wu Y., Zeng J., Grove J., Kim M., Li Z., Voloudakis G., Zhang W., Adams M., Agartz I., Atkinson E.G., Agerbo E., Al Eissa M., Albus M., Alexander M., Alizadeh B.Z., Alptekin K., Als T.D., Amin F., Arolt V., Arrojo M., Athanasiu L., Azevedo M.H., Bacanu S.A., Bass N.J., Begemann M., Belliveau R.A., Bene J., Benyamin B., Bergen S.E., Blasi G., Bobes J., Bonassi S., Braun A., Bressan R.A., Bromet E.J., Bruggeman R., Buckley P.F., Buckner R.L., Bybjerg-Grauholm J., Cahn W., Cairns M.J., Calkins M.E., Carr V.J., Castle D., Catts S.V., Chambert K.D., Chan R.C.K., Chaumette B., Cheng W., Cheung E.F.C., Chong S.A., Cohen D., Consoli A., Cordeiro Q., Costas J., Curtis C., Davidson M., Davis K.L., de Haan L., Degenhardt F., DeLisi L.E., Demontis D., Dickerson F., Dikeos D., Dinan T., Djurovic S., Duan J., Ducci G., Dudbridge F., Eriksson J.G., Fañanás L., Faraone S.V., Fiorentino A., Forstner A., Frank J., Freimer N.B., Fromer M., Frustaci A., Gadelha A., Genovese G., Gershon E.S., Giannitelli M., Giegling I., Giusti-Rodríguez P., Godard S., Goldstein J.I., González Peñas J., González-Pinto A., Gopal S., Gratten J., Green M.F., Greenwood T.A., Guillin O., Gülöksüz S., Gur R.E., Gur R.C., Gutiérrez B., Hahn E., Hakonarson H., Haroutunian V., Hartmann A.M., Harvey C., Hayward C., Henskens F.A., Herms S., Hoffmann P., Howrigan D.P., Ikeda M., Iyegbe C., Joa I., Julià A., Kähler A.K., Kam-Thong T., Kamatani Y., Karachanak-Yankova S., Kebir O., Keller M.C., Kelly B.J., Khrunin A., Kim S.W., Klovins J., Kondratiev N., Konte B., Kraft J., Kubo M., Kučinskas V., Kučinskiene Z.A., Kusumawardhani A., Kuzelova-Ptackova H., Landi S., Lazzeroni L.C., Lee P.H., Legge S.E., Lehrer D.S., Lencer R., Lerer B., Li M., Lieberman J., Light G.A., Limborska S., Liu C.M., Lönnqvist J., Loughland C.M., Lubinski J., Luykx J.J., Lynham A., Macek M. Jr, Mackinnon A., Magnusson P.K.E., Maher B.S., Maier W., Malaspina D., Mallet J., Marder S.R., Marsal S., Martin A.R., Martorell L., Mattheisen M., McCarley R.W., McDonald C., McGrath J.J., Medeiros H., Meier S., Melegh B., Melle I., Mesholam-Gately R.I., Metspalu A., Michie P.T., Milani L., Milanova V., Mitjans M., Molden E., Molina E., Molto M.D., Mondelli V., Moreno C., Morley C.P., Muntané G., Murphy K.C., Myin-Germeys I., Nenadić I., Nestadt G., Nikitina-Zake L., Noto C., Nuechterlein K.H., O’Brien N.L., O’Neill F.A., Oh S.Y., Olincy A., Ota V.K., Pantelis C., Papadimitriou G.N., Parellada M., Paunio T., Pellegrino R., Periyasamy S., Perkins D.O., Pfuhlmann B., Pietiläinen O., Pimm J., Porteous D., Powell J., Quattrone D., Quested D., Radant A.D., Rampino A., Rapaport M.H., Rautanen A., Reichenberg A., Roe C., Roffman J.L., Roth J., Rothermundt M., Rutten B.P.F., Saker-Delye S., Salomaa V., Sanjuan J., Santoro M.L., Savitz A., Schall U., Scott R.J., Seidman L.J., Sharp S.I., Shi J., Siever L.J., Sigurdsson E., Sim K., Skarabis N., Slominsky P., So H.C., Sobell J.L., Söderman E., Stain H.J., Steen N.E., Steixner-Kumar A.A., Stögmann E., Stone W.S., Straub R.E., Streit F., Strengman E., Stroup T.S., Subramaniam M., Sugar C.A., Suvisaari J., Svrakic D.M., Swerdlow N.R., Szatkiewicz J.P., Ta T.M.T., Takahashi A., Terao C., Thibaut F., Toncheva D., Tooney P.A., Torretta S., Tosato S., Tura G.B., Turetsky B.I., Üçok A., Vaaler A., van Amelsvoort T., van Winkel R., Veijola J., Waddington J., Walter H., Waterreus A., Webb B.T., Weiser M., Williams N.M., Witt S.H., Wormley B.K., Wu J.Q., Xu Z., Yolken R., Zai C.C., Zhou W., Zhu F., Zimprich F., Atbaşoğlu E.C., Ayub M., Benner C., Bertolino A., Black D.W., Bray N.J., Breen G., Buccola N.G., Byerley W.F., Chen W.J., Cloninger C.R., Crespo-Facorro B., Donohoe G., Freedman R., Galletly C., Gandal M.J., Gennarelli M., Hougaard D.M., Hwu H.G., Jablensky A.V., McCarroll S.A., Moran J.L., Mors O., Mortensen P.B., Müller-Myhsok B., Neil A.L., Nordentoft M., Pato M.T., Petryshen T.L., Pirinen M., Pulver A.E., Schulze T.G., Silverman J.M., Smoller J.W., Stahl E.A., Tsuang D.W., Vilella E., Wang S.H., Xu S.; Indonesia Schizophrenia Consortium; PsychENCODE; Psychosis Endophenotypes International Consortium; SynGO Consortium; Adolfsson R., Arango C., Baune B.T., Belangero S.I., Børglum A.D., Braff D., Bramon E., Buxbaum J.D., Campion D., Cervilla J.A., Cichon S., Collier D.A., Corvin A., Curtis D., Forti M.D., Domenici E., Ehrenreich H., Escott-Price V., Esko T., Fanous A.H., Gareeva A., Gawlik M., Gejman P.V., Gill M., Glatt S.J., Golimbet V., Hong K.S., Hultman C.M., Hyman S.E., Iwata N., Jönsson E.G., Kahn R.S., Kennedy J.L., Khusnutdinova E., Kirov G., Knowles J.A., Krebs M.O., Laurent-Levinson C., Lee J., Lencz T., Levinson D.F., Li Q.S., Liu J., Malhotra A.K., Malhotra D., McIntosh A., McQuillin A., Menezes P.R., Morgan V.A., Morris D.W., Mowry B.J., Murray R.M., Nimgaonkar V., Nöthen M.M., Ophoff R.A., Paciga S.A., Palotie A., Pato C.N., Qin S., Rietschel M., Riley B.P., Rivera M., Rujescu D., Saka M.C., Sanders A.R., Schwab S.G., Serretti A., Sham P.C., Shi Y., St. Clair D., Stefánsson H., Stefansson K., Tsuang M.T., van Os J., Vawter M.P., Weinberger D.R., Werge T., Wildenauer D.B., Yu X., Yue W., Holmans P.A., Pocklington A.J., Roussos P., Vassos E., Verhage M., Visscher P.M., Yang J., Posthuma D., Andreassen O.A., Kendler K.S., Owen M.J., Wray N.R., Daly M.J., Huang H., Neale B.M., Sullivan P.F., Ripke S., Walters J.T.R., O’Donovan M.C.; Schizophrenia Working Group of the Psychiatric Genomics Consortium. Mapping genomic loci implicates genes and synaptic biology in schizophrenia. Nature 2022; 604(7906): 502–508, https://doi.org/10.1038/s41586-022-04434-5.
- Sullivan P.F., Yao S., Hjerling-Leffler J. Schizophrenia genomics: genetic complexity and functional insights. Nat Rev Neurosci 2024; 25(9): 611–624, https://doi.org/10.1038/s41583-024-00837-7.
- Singh T., Poterba T., Curtis D., Akil H., Al Eissa M., Barchas J.D., Bass N., Bigdeli T.B., Breen G., Bromet E.J., Buckley P.F., Bunney W.E., Bybjerg-Grauholm J., Byerley W.F., Chapman S.B., Chen W.J., Churchhouse C., Craddock N., Cusick C.M., DeLisi L., Dodge S., Escamilla M.A., Eskelinen S., Fanous A.H., Faraone S.V., Fiorentino A., Francioli L., Gabriel S.B., Gage D., Gagliano Taliun S.A., Ganna A., Genovese G., Glahn D.C., Grove J., Hall M.H., Hämäläinen E., Heyne H.O., Holi M., Hougaard D.M., Howrigan D.P., Huang H., Hwu H.G., Kahn R.S., Kang H.M., Karczewski K.J., Kirov G., Knowles J.A., Lee F.S., Lehrer D.S., Lescai F., Malaspina D., Marder S.R., McCarroll S.A., McIntosh A.M., Medeiros H., Milani L., Morley C.P., Morris D.W., Mortensen P.B., Myers R.M., Nordentoft M., O’Brien N.L., Olivares A.M., Ongur D., Ouwehand W.H., Palmer D.S., Paunio T., Quested D., Rapaport M.H., Rees E., Rollins B., Satterstrom F.K., Schatzberg A., Scolnick E., Scott L.J., Sharp S.I., Sklar P., Smoller J.W., Sobell J.L., Solomonson M., Stahl E.A., Stevens C.R., Suvisaari J., Tiao G., Watson S.J., Watts N.A., Blackwood D.H., Børglum A.D., Cohen B.M., Corvin A.P., Esko T., Freimer N.B., Glatt S.J., Hultman C.M., McQuillin A., Palotie A., Pato C.N., Pato M.T., Pulver A.E., St. Clair D., Tsuang M.T., Vawter M.P., Walters J.T., Werge T.M., Ophoff R.A., Sullivan P.F., Owen M.J., Boehnke M., O’Donovan M.C., Neale B.M., Daly M.J. Rare coding variants in ten genes confer substantial risk for schizophrenia. Nature 2022; 604(7906): 509–516, https://doi.org/10.1038/s41586-022-04556-w.
- Leger B.S., Meredith J.J., Ideker T., Sanchez-Roige S., Palmer A.A. Rare and common variants associated with alcohol consumption identify a conserved molecular network. Alcohol Clin Exp Res (Hoboken) 2024; 48(9): 1704–1715, https://doi.org/10.1111/acer.15399.
- Ezewudo M., Zwick M.E. Evaluating rare variants in complex disorders using next-generation sequencing. Curr Psychiatry Rep 2013; 15(4): 349, https://doi.org/10.1007/s11920-013-0349-4.
- Schreiber M., Dorschner M., Tsuang D. Next-generation sequencing in schizophrenia and other neuropsychiatric disorders. Am J Med Genet B Neuropsychiatr Genet 2013; 162B(7): 671–678, https://doi.org/10.1002/ajmg.b.32156.
- Ithal D., Sukumaran S.K., Bhattacharjee D., Vemula A., Nadella R., Mahadevan J., Sud R., Viswanath B., Purushottam M., Jain S. Exome hits demystified: the next frontier. Asian J Psychiatr 2021; 59: 102640, https://doi.org/10.1016/j.ajp.2021.102640.
- Kato H., Kimura H., Kushima I., Takahashi N., Aleksic B., Ozaki N. The genetic architecture of schizophrenia: review of large-scale genetic studies. J Hum Genet 2023; 68(3): 175–182, https://doi.org/10.1038/s10038-022-01059-4.
- Pouget J.G. The emerging immunogenetic architecture of schizophrenia. Schizophr Bull 2018; 44(5): 993–1004, https://doi.org/10.1093/schbul/sby038.
- Малашенкова И.К., Крынский С.А., Огурцов Д.П., Мамошина М.В., Захарова Н.В., Ушаков В.Л., Величковский Б.М., Дидковский Н.А. Роль иммунной системы в патогенезе шизофрении. Журнал неврологии и психиатрии им. С.С. Корсакова 2018; 118(12): 72–80, https://doi.org/10.17116/jnevro201811812172.
- Mikhalitskaya E.V., Vyalova N.M., Ermakov E.A., Levchuk L.A., Simutkin G.G., Bokhan N.A., Ivanova S.A. Association of single nucleotide polymorphisms of cytokine genes with depression, schizophrenia and bipolar disorder. Genes (Basel) 2023; 14(7): 1460, https://doi.org/ 10.3390/genes14071460.
- Malashenkova I.K., Ushakov V.L., Krynskiy S.A., Ogurtsov D.P., Khailov N.A., Ratushnyy A.Yu., Filippova E.A., Zakharova N.V., Kostyuk G.P., Didkovsky N.A. Associations of IL17A G-197A single nucleotide polymorphism with immunological parameters and structural changes of the brain in schizophrenia. Medical Immunology (Russia) 2023; 25(5): 1225–1232, https://doi.org/10.15789/1563-0625-aoi-2806.
- Malashenkova I.K., Ushakov V.L., Krynskiy S.A., Ogurtsov D.P., Chekulaeva E.I., Filippova E.A., Orlov V.A., Zakharova N.V., Andreyuk D.S., Trushchelev S.A., Kostyuk G.P., Didkovsky N.A. Polymorphisms of IL10 immunoregulatory gene impact the morphometric changes of the brain in schizophrenia. In: Samsonovich A.V., Liu T. (editors). Biologically inspired cognitive architectures 2023. BICA 2023. Studies in Computational Intelligence, vol. 1130. Springer, Cham; 2024; p. 577–587, https://doi.org/10.1007/978-3-031-50381-8_61.
- Morozova A., Zorkina Y., Pavlov K., Pavlova O., Abramova O., Ushakova V., Mudrak A.V., Zozulya S., Otman I., Sarmanova Z., Klyushnik T., Reznik A., Kostyuk G., Chekhonin V. Associations of genetic polymorphisms and neuroimmune markers with some parameters of frontal lobe dysfunction in schizophrenia. Front Psychiatry 2021; 12: 655178, https://doi.org/10.3389/fpsyt.2021.655178.
- Duan L., Li S., Chen D., Shi Y., Zhou X., Feng Y. Causality between autoimmune diseases and schizophrenia: a bidirectional Mendelian randomization study. BMC Psychiatry 2024; 24(1): 817, https://doi.org/10.1186/s12888-024-06287-w.
- Wang R., Lu J.Y., Herbert D., Lieberman J.A., Meltzer H.Y., Tiwari A.K., Remington G., Kennedy J.L., Zai C.C. Analysis of schizophrenia-associated genetic markers in the HLA region as risk factors for tardive dyskinesia. Hum Psychopharmacol 2024; 39(4): e2898, https://doi.org/10.1002/hup.2898.
- Langmead B., Wilks C., Antonescu V., Charles R. Scaling read aligners to hundreds of threads on general-purpose processors. Bioinformatics 2019; 35(3): 421–432, https://doi.org/10.1093/bioinformatics/bty648.
- Martin F.J., Amode M.R., Aneja A., Austine-Orimoloye O., Azov A.G., Barnes I., Becker A., Bennett R., Berry A., Bhai J., Bhurji S.K., Bignell A., Boddu S., Branco Lins P.R., Brooks L., Ramaraju S.B., Charkhchi M., Cockburn A., Da Rin Fiorretto L., Davidson C., Dodiya K., Donaldson S., El Houdaigui B., El Naboulsi T., Fatima R., Giron C.G., Genez T., Ghattaoraya G.S., Martinez J.G., Guijarro C., Hardy M., Hollis Z., Hourlier T., Hunt T., Kay M., Kaykala V., Le T., Lemos D., Marques-Coelho D., Marugán J.C., Merino G.A., Mirabueno L.P., Mushtaq A., Hossain S.N., Ogeh D.N., Sakthivel M.P., Parker A., Perry M., Piližota I., Prosovetskaia I., Pérez-Silva J.G., Salam A.I.A., Saraiva-Agostinho N., Schuilenburg H., Sheppard D., Sinha S., Sipos B., Stark W., Steed E., Sukumaran R., Sumathipala D., Suner M.M., Surapaneni L., Sutinen K., Szpak M., Tricomi F.F., Urbina-Gómez D., Veidenberg A., Walsh T.A., Walts B., Wass E., Willhoft N., Allen J., Alvarez-Jarreta J., Chakiachvili M., Flint B., Giorgetti S., Haggerty L., Ilsley G.R., Loveland J.E., Moore B., Mudge J.M., Tate J., Thybert D., Trevanion S.J., Winterbottom A., Frankish A., Hunt S.E., Ruffier M., Cunningham F., Dyer S., Finn R.D., Howe K.L., Harrison P.W., Yates A.D., Flicek P. Ensembl 2023. Nucleic Acids Res 2023; 51(D1): D933–D941, https://doi.org/10.1093/nar/gkac958.
- Quinlan A.R., Hall I.M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics 2010; 26(6): 841–842, https://doi.org/10.1093/bioinformatics/btq033.
- Li H., Handsaker B., Wysoker A., Fennell T., Ruan J., Homer N., Marth G., Abecasis G., Durbin R.; 1000 Genome Project Data Processing Subgroup. the sequence alignment/Map format and SAMtools. Bioinformatics 2009; 25(16): 2078–2079, https://doi.org/10.1093/bioinformatics/btp352.
- Li H. A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data. Bioinformatics 2011; 27(21): 2987–2993, https://doi.org/10.1093/bioinformatics/btr509.
- Vaser R., Adusumalli S., Leng S.N., Sikic M., Ng P.C. SIFT missense predictions for genomes. Nat Protoc 2016; 11(1): 1–9, https://doi.org/10.1038/nprot.2015.123.
- Sherman B.T., Hao M., Qiu J., Jiao X., Baseler M.W., Lane H.C., Imamichi T., Chang W. DAVID: a web server for functional enrichment analysis and functional annotation of gene lists (2021 update). Nucleic Acids Res 2022; 50(W1): W216–W221, https://doi.org/10.1093/nar/gkac194.
- Zurlo L., Dal Bò E., Gentili C., Cecchetto C. Olfactory dysfunction in schizophrenia and other psychotic disorders: a comprehensive and updated meta-analysis. Schizophr Res 2025; 275: 62–75, https://doi.org/10.1016/j.schres.2024.12.001.
- Морозова С.В., Савватеева Д.М., Тимурзиева А.Б. Обонятельные расстройства у пациентов с психическими заболеваниями. Журнал неврологии и психиатрии им. С.С. Корсакова 2014; 114(7): 73–78.
- Turetsky B.I., Moberg P.J., Quarmley M., Dress E., Calkins M.E., Ruparel K., Prabhakaran K., Gur R.E., Roalf D.R. Structural anomalies of the peripheral olfactory system in psychosis high-risk subjects. Schizophr Res 2018; 195: 197–205, https://doi.org/10.1016/j.schres.2017.09.015.
- Ullah M.N., Rowan N.R., Lane A.P. Neuroimmune interactions in the olfactory epithelium: maintaining a sensory organ at an immune barrier interface. Trends Immunol 2024; 45(12): 987–1000, https://doi.org/10.1016/j.it.2024.10.005.
- Lenormand C., Bausinger H., Gross F., Signorino-Gelo F., Koch S., Peressin M., Fricker D., Cazenave J.P., Bieber T., Hanau D., de la Salle H., Tourne S. HLA-DQA2 and HLA-DQB2 genes are specifically expressed in human Langerhans cells and encode a new HLA class II molecule. J Immunol 2012; 188(8): 3903–3911, https://doi.org/10.4049/jimmunol.1103048.
- Tamouza R., Krishnamoorthy R., Leboyer M. Understanding the genetic contribution of the human leukocyte antigen system to common major psychiatric disorders in a world pandemic context. Brain Behav Immun 2021; 91: 731–739, https://doi.org/10.1016/j.bbi.2020.09.033.
- Mine K.L., Tedesco-Silva H., Mourão T.B., Campos E.F., Salzedas L.A., Aguiar B., Felipe C.R., Medina-Pestana J.O., Gerbase-DeLima M. Heightened expression of HLA-DQB1 and HLA-DQB2 in pre-implantation biopsies predicts poor late kidney graft function. Hum Immunol 2018; 79(8): 594–601, https://doi.org/10.1016/j.humimm.2018.05.002.
- Mine K.L., de Marco R., Raimundo T.R.F., Ernesto J.V., Medina-Pestana J.O., Tedesco-Silva H., Gerbase-DeLima M. High soluble HLA-DQB2 levels in posttransplant serum are associated with kidney graft dysfunction. Int J Immunogenet 2022; 49(2): 63–69, https://doi.org/10.1111/iji.12569.
- Wu G., Xiao G., Yan Y., Guo C., Hu N., Shen S. Bioinformatics analysis of the clinical significance of HLA class II in breast cancer. Medicine (Baltimore) 2022; 101(40): e31071, https://doi.org/10.1097/MD.0000000000031071.
- Ureña-Guerrero M.E., Castañeda-Cabral J.L., Rivera-Cervantes M.C., Macias-Velez R.J., Jarero-Basulto J.J., Gudiño-Cabrera G., Beas-Zárate C. Neuroprotective and neurorestorative effects of Epo and VEGF: perspectives for new therapeutic approaches to neurological diseases. Curr Pharm Des 2020; 26(12): 1263–1276, https://doi.org/10.2174/1381612826666200114104342.
- Pillai A., Howell K.R., Ahmed A.O., Weinberg D., Allen K.M., Bruggemann J., Lenroot R., Liu D., Galletly C., Weickert C.S., Weickert T.W. Association of serum VEGF levels with prefrontal cortex volume in schizophrenia. Mol Psychiatry 2016; 21(5): 686–692, https://doi.org/10.1038/mp.2015.96.
- Malashenkova I.K., Ushakov V.L., Krynskiy S.A., Ogurtsov D.P., Khailov N.A., Chekulaeva E.I., Filippova E.A., Orlov V.A., Didkovsky N.A., Zakharova N.V., Andreyuk D.A., Kostyuk G.P. Associations of morphometric changes of the brain with the levels of IGF1, a multifunctional growth factor, and with systemic immune parameters reflect the disturbances of neuroimmune interactions in patients with schizophrenia. In: Kryzhanovsky B., Dunin-Barkowski W., Redko V., Tiumentsev Y. (editors). Advances in neural computation, machine learning, and cognitive research VI. Cham: Springer International Publishing; 2023; p. 167–179, https://doi.org/10.1007/978-3-031-19032-2_17.
- Yesilkaya U.H., Gica S., Ilnem M.C., Sen M., Ipekcioglu D. Evaluation of IGF-1 as a novel theranostic biomarker for schizophrenia. J Psychiatr Res 2021; 140: 172–179, https://doi.org/10.1016/j.jpsychires.2021.05.078.
- Allen N.C., Bagade S., McQueen M.B., Ioannidis J.P., Kavvoura F.K., Khoury M.J., Tanzi R.E., Bertram L. Systematic meta-analyses and field synopsis of genetic association studies in schizophrenia: the SzGene database. Nat Genet 2008; 40(7): 827–834, https://doi.org/10.1038/ng.171.
- Jia P., Han G., Zhao J., Lu P., Zhao Z. SZGR 2.0: a one-stop shop of schizophrenia candidate genes. Nucleic Acids Res 2017; 45(D1): D915–D924, https://doi.org/ 10.1093/nar/gkw902.
- Wu Y., Yao Y.G., Luo X.J. SZDB: a database for schizophrenia genetic research. Schizophr Bull 2017; 43(2): 459–471, https://doi.org/10.1093/schbul/sbw102.
- Li J., Yoshikawa A., Alliey-Rodriguez N., Meltzer H.Y. Schizophrenia risk loci from xMHC region were associated with antipsychotic response in chronic schizophrenic patients with persistent positive symptom. Transl Psychiatry 2022; 12(1): 92, https://doi.org/10.1038/s41398-022-01854-9.
- Islam F., Hain D., Lewis D., Law R., Brown L.C., Tanner J.A., Müller D.J. Pharmacogenomics of Clozapine-induced agranulocytosis: a systematic review and meta-analysis. Pharmacogenomics J 2022; 22(4): 230–240, https://doi.org/10.1038/s41397-022-00281-9.
- Seshasubramanian V., Raghavan V., SathishKannan A.D., Naganathan C., Ramachandran A., Arasu P., Rajendren P., John S., Mowry B., Rangaswamy T., Narayan S., Periathiruvadi S. Association of HLA-A, -B, -C, -DRB1 and -DQB1 alleles at amino acid level in individuals with schizophrenia: a study from South India. Int J Immunogenet 2020; 47(6): 501–511, https://doi.org/10.1111/iji.12507.
- Katrinli S., Lori A., Kilaru V., Carter S., Powers A., Gillespie C.F., Wingo A.P., Michopoulos V., Jovanovic T., Ressler K.J., Smith A.K. Association of HLA locus alleles with posttraumatic stress disorder. Brain Behav Immun 2019; 81: 655–658, https://doi.org/10.1016/j.bbi.2019.07.016.
- Smith K.R., Penzes P. Ankyrins: roles in synaptic biology and pathology. Mol Cell Neurosci 2018; 91: 131–139, https://doi.org/ 10.1016/j.mcn.2018.04.010.
- He F., Zhou Y.M., Qi Y.J., Huang H.H., Guan L., Luo J., Cheng Y.H., Zheng Y. Exploration of mutated genes and prediction of potential biomarkers for childhood-onset schizophrenia using an integrated bioinformatic analysis. Front Aging Neurosci 2022; 14: 829217, https://doi.org/10.3389/fnagi.2022.829217.
- Chen J., Zhao X., Cui L., He G., Wang X., Wang F., Duan S., He L., Li Q., Yu X., Zhang F., Xu M. Genetic regulatory subnetworks and key regulating genes in rat hippocampus perturbed by prenatal malnutrition: implications for major brain disorders. Aging (Albany NY) 2020; 12(9): 8434–8458, https://doi.org/10.18632/aging.103150.










